python 상관계수 그래프 그리기
python 상관계수 그래프를 그리는 방법을 상세하게 소개하려고 합니다.
해당 코드를 실습할수 있는 데이터는
캐글 데이터 페이지를 통해서 다운로드 부탁드리겠습니다.
import warnings
warnings.filterwarnings(action='ignore')
import numpy as np
import pandas as pd
import seaborn as sns
from operator import itemgetter
%matplotlib inline
import matplotlib.pyplot as plt
from matplotlib.patches import Rectangle
data = pd.read_csv("2019_kbo_for_kaggle_v2.csv")
print(data.shape)
data.head()
(1913, 37)
| batter_name | age | G | PA | AB | R | H | 2B | 3B | HR | ... | tp | 1B | FBP | avg | OBP | SLG | OPS | p_year | YAB | YOPS | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 백용환 | 24.0 | 26.0 | 58.0 | 52.0 | 4.0 | 9.0 | 4.0 | 0.0 | 0.0 | ... | 포수 | 5.0 | 6.0 | 0.173 | 0.259 | 0.250 | 0.509 | 2014 | 79.0 | 0.580 |
| 1 | 백용환 | 25.0 | 47.0 | 86.0 | 79.0 | 8.0 | 14.0 | 2.0 | 0.0 | 4.0 | ... | 포수 | 8.0 | 5.0 | 0.177 | 0.226 | 0.354 | 0.580 | 2015 | 154.0 | 0.784 |
| 2 | 백용환 | 26.0 | 65.0 | 177.0 | 154.0 | 22.0 | 36.0 | 6.0 | 0.0 | 10.0 | ... | 포수 | 20.0 | 20.0 | 0.234 | 0.316 | 0.468 | 0.784 | 2016 | 174.0 | 0.581 |
| 3 | 백용환 | 27.0 | 80.0 | 199.0 | 174.0 | 12.0 | 34.0 | 7.0 | 0.0 | 4.0 | ... | 포수 | 23.0 | 20.0 | 0.195 | 0.276 | 0.305 | 0.581 | 2017 | 17.0 | 0.476 |
| 4 | 백용환 | 28.0 | 15.0 | 20.0 | 17.0 | 2.0 | 3.0 | 0.0 | 0.0 | 0.0 | ... | 포수 | 3.0 | 3.0 | 0.176 | 0.300 | 0.176 | 0.476 | 2018 | 47.0 | 0.691 |
5 rows × 37 columns
추가 데이터 처리 후 corr 그래프
데이터의 컬럼이 많아 데이터 중 일부 컬럼만 사용하도록 하겠습니다.
regular_y2 = data.copy()
regular_y2 = regular_y2[['age', 'AB', 'HR',
'BB', 'SO', 'avg', 'OPS', 'YAB', 'YOPS']]
corrlation 그래프는 seaborn에서의 heatmap을 통해서 그릴 수 있는데
먼저 데이터에 상관계수 값을 구해줍니다.
cor = regular_y2.corr()
cor
| age | AB | HR | BB | SO | avg | OPS | YAB | YOPS | |
|---|---|---|---|---|---|---|---|---|---|
| age | 1.000000 | 0.295713 | 0.265501 | 0.298501 | 0.255230 | 0.197390 | 0.227824 | 0.152473 | 0.173237 |
| AB | 0.295713 | 1.000000 | 0.708748 | 0.872795 | 0.871873 | 0.489783 | 0.542863 | 0.719471 | 0.479462 |
| HR | 0.265501 | 0.708748 | 1.000000 | 0.737227 | 0.751261 | 0.366961 | 0.540203 | 0.561271 | 0.471787 |
| BB | 0.298501 | 0.872795 | 0.737227 | 1.000000 | 0.789912 | 0.441878 | 0.547978 | 0.640951 | 0.480934 |
| SO | 0.255230 | 0.871873 | 0.751261 | 0.789912 | 1.000000 | 0.400740 | 0.506455 | 0.610473 | 0.435305 |
| avg | 0.197390 | 0.489783 | 0.366961 | 0.441878 | 0.400740 | 1.000000 | 0.923942 | 0.409053 | 0.344919 |
| OPS | 0.227824 | 0.542863 | 0.540203 | 0.547978 | 0.506455 | 0.923942 | 1.000000 | 0.464115 | 0.412618 |
| YAB | 0.152473 | 0.719471 | 0.561271 | 0.640951 | 0.610473 | 0.409053 | 0.464115 | 1.000000 | 0.616505 |
| YOPS | 0.173237 | 0.479462 | 0.471787 | 0.480934 | 0.435305 | 0.344919 | 0.412618 | 0.616505 | 1.000000 |
상관계수 값을 구한 다음에는 heatmap에 cor 값을 넣으면 자동으로 default 설정에 따라 그려집니다.
여기서 annot는 그래프 칸 하나 하나에 숫자를 입력해주는 것을 할 것인지에 대한 것입니다.
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
sns.heatmap(cor, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0);
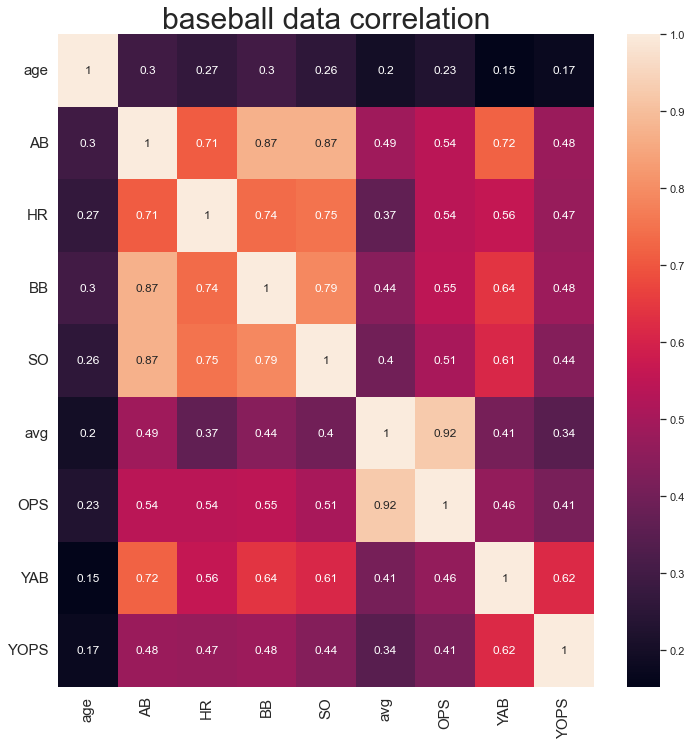
위의 코드에서는 기본 설정만으로 그리고, plt.subplots를 통해서 x축을 90도 회전 해보았습니다.
다만 세로가 길어보이는 느낌이 들고 색깔이 중구 난방이라 수정을 해주고 싶어집니다.
아래의 코드에서는 색깔 기준점인 center를 0.5에 두고, square=True를 통해서 정사각형 모양으로 바꿨습니다.
그리고 linewidths=0.5를 통해서 각 사각형 마다 흰 색 선을 추가해 줬으며,
cbar_kws={“shrink”: 0.75}를 통해서 오른쪽 막대의 크기를 살짝 줄여주었습니다.
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
sns.heatmap(cor, center=0.5, square=True,
linewidths=0.5, cbar_kws={"shrink": 0.75}, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0);
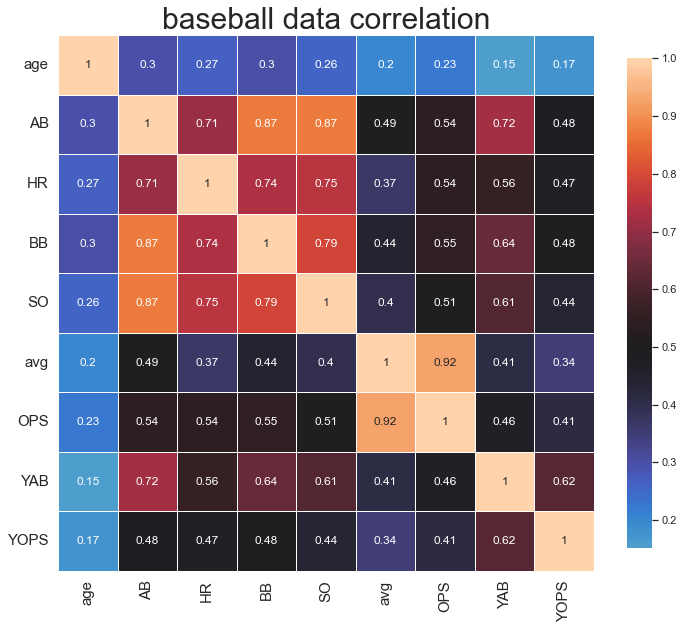
중심을 0.5로 두니 값이 0.5에 가까우면 검정색,
1에 가까울수록 빨간색쪽으로
0에 가까울수록 파란색쪽으로 나타나게 됩니다.
하지만 이를 검정색이 아닌 흰색으로 하고 싶을때는 아래의 코드를 활용해줘야 합니다.
heatmap에서 cmap이라는 항목에 해당 객체를 추가하게 된다면
center 값이 검정색이 아닌 흰색으로 지정이 됩니다.
cmap = sns.diverging_palette(200, 10, as_cmap=True)
cmap
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
cmap = sns.diverging_palette(200, 10, as_cmap=True)
sns.heatmap(cor, cmap=cmap, center=0.5,
square=True, linewidths=0.5, cbar_kws={"shrink": 0.75}, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0);
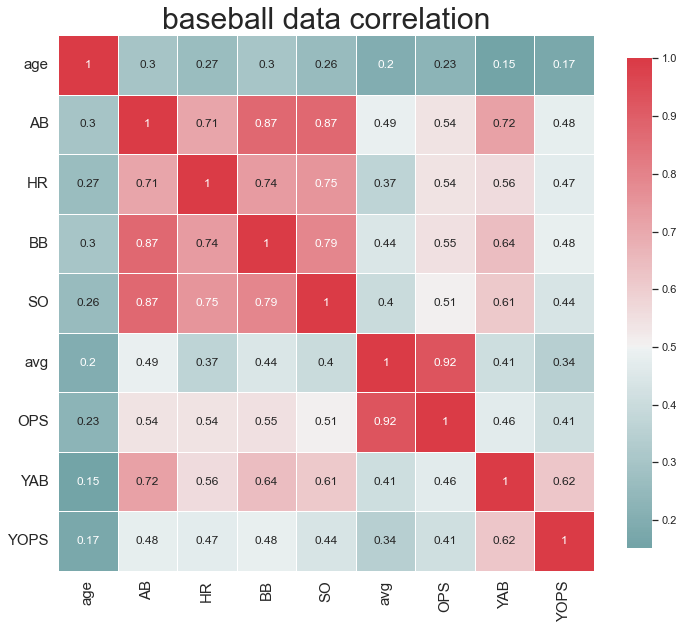
cmap에서 확인한 색깔 설정처럼 0.5에 가까우면 흰색이 되고
전보다는 색깔이 파스텔 톤으로 바뀐 것을 확인할 수 있습니다.
center를 0으로 설정하게 된다면 아래와 같이 모든 값이 0보다 크기 때문에
빨간색으로만 명암이 구분되고 그래프가 그려지게 됩니다
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
cmap = sns.diverging_palette(200, 10, as_cmap=True)
sns.heatmap(cor, cmap=cmap, center=0,
square=True, linewidths=0.5, cbar_kws={"shrink": 0.75}, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0);
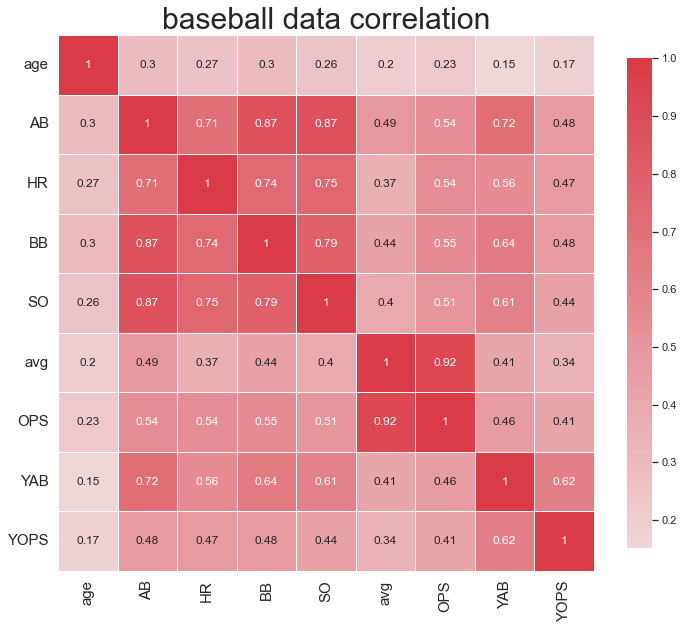
위에 코드에서는 기본적인 설정에서 코드 수정을 통해 그래프 자체의 가시성을 개선하고
색깔을 자유롭게 바꾸고 색깔의 기준점을 자유롭게 설정하는 방법을 소개해드렸습니다.
위 그림도 좋을수 있지만, 저는 개인적으로 같은 변수일때 나타나는 1의 값과
윗 부분과 아래 부분의 값이 같은 것 특성 때문에 아래 부분만 보여주는 것을
코드를 통해서 추가로 구현하고자 합니다.
먼저 correlation 값을 저장한 객체를 통해서 같은 크기의 False array를 만들어냅니다.
mask = np.zeros_like(cor, dtype=np.bool)
mask
array([[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False],
[False, False, False, False, False, False, False, False, False]])
그리고 대각선 영역과 대각성 영역 윗 부분에 True를 설정해줍니다.
mask[np.triu_indices_from(mask)] = True
mask
array([[ True, True, True, True, True, True, True, True, True],
[False, True, True, True, True, True, True, True, True],
[False, False, True, True, True, True, True, True, True],
[False, False, False, True, True, True, True, True, True],
[False, False, False, False, True, True, True, True, True],
[False, False, False, False, False, True, True, True, True],
[False, False, False, False, False, False, True, True, True],
[False, False, False, False, False, False, False, True, True],
[False, False, False, False, False, False, False, False, True]])
그리고 이를 heatmap안에서 mask안에 넣어주면 False 영역만 나타나게 됩니다.
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
cmap = sns.diverging_palette(200, 10, as_cmap=True)
mask = np.zeros_like(cor, dtype=np.bool)
mask[np.triu_indices_from(mask)] = True
sns.heatmap(cor, mask=mask, cmap=cmap, center=0.5, square=True,
linewidths=0.5, cbar_kws={"shrink": 0.75}, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0);
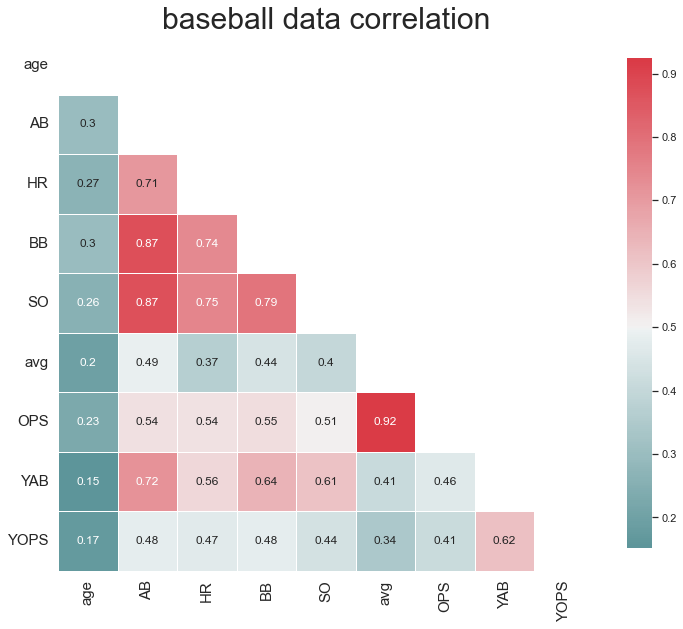
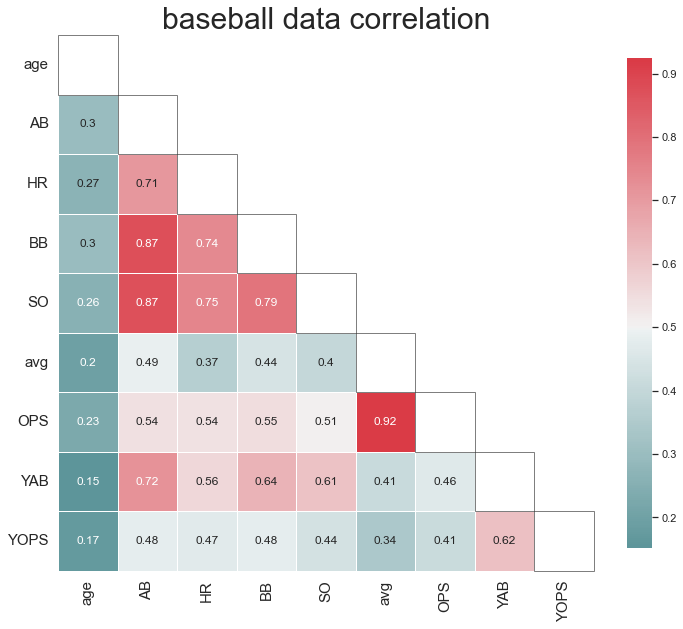
이렇게 마무리를 해도 좋지만, 이렇게 대각선 영역을 빼고 그리게 되면
변수가 많이 존재할때에는 변수들을 찾아가면서 보기가 힘든 경우가 생겼습니다.
그렇기에 마지막 영역에 plt의 Rectangle를 통해서 각 대각선 마다 작은 사각형을 추가해 주었습니다.
sns.set(style="white")
cor = regular_y2.corr()
f, ax = plt.subplots(figsize=(12, 12))
cmap = sns.diverging_palette(200, 10, as_cmap=True)
mask = np.zeros_like(cor, dtype=np.bool)
mask[np.triu_indices_from(mask)] = True
sns.heatmap(cor, mask=mask, cmap=cmap, center=0.5, square=True,
linewidths=0.5, cbar_kws={"shrink": 0.75}, annot=True)
plt.title('baseball data correlation', size=30)
ax.set_xticklabels(list(regular_y2.columns), size=15, rotation=90)
ax.set_yticklabels(list(regular_y2.columns), size=15, rotation=0)
for temp_num in range(len(regular_y2.columns)):
ax.add_patch(Rectangle((temp_num, temp_num), 1, 1, fill=False,
edgecolor='black', lw=1, clip_on=False, alpha=0.5))